|
|
АкушерствоАнатомияАнестезиологияВакцинопрофилактикаВалеологияВетеринарияГигиенаЗаболеванияИммунологияКардиологияНеврологияНефрологияОнкологияОториноларингологияОфтальмологияПаразитологияПедиатрияПервая помощьПсихиатрияПульмонологияРеанимацияРевматологияСтоматологияТерапияТоксикологияТравматологияУрологияФармакологияФармацевтикаФизиотерапияФтизиатрияХирургияЭндокринологияЭпидемиология |
Масштабированные и немасштабированные дендрограммы
Компоненты эволюционных деревьев (рис. 18): Корень – соответствует общему предшественнику всех таксонов. Узел – место соединения ветвей, объединяющее представителей одной таксономи-ческой группы (одного вида, однотипные штаммы). Ветвь – графическое изображение взаимосвязи между таксонами. Длина ветви может отражать количество изменений, произошедших в данном ответвлении. Топология – характер расположения ветвей. Клады – группа из двух и более таксонов, включающая общего предшественника и все произошедшие от него монофилетические группы (все родственныепоследовательности ДНК, произошедшие от одной общей ДНК-предшественника). Изучение эволюции генов и белков требует проведения сравнений между гомологичными последовательностями ДНК/протеинов. Существуют следующие варианты гомологичных генов: Ортологи – гомологичные гены, произошедшие от общего гена-предшественника в ходе естественного видообразования. Несмотря на некоторые структурные отличия, ортологи имеют сходные функции. Паралоги – гомологичные гены, произошедшие в результате удвоения гена-предшественника. Удвоившиеся гены одного организма претерпевают независимые изменения, вследствие чего нередко обладают разными функциями. Ксенологи – гомологичные гены, произошедшие в результате горизонтального переноса генов между организмами. Функции могут быть как идентичными, так и разными.
Литература 1. Методы общей бактериологии: Пер. с англ./ Под ред. Герхарда Ф. и др. – М.: Мир, 1984. – 472 с. 2. Молекулярная клиническая диагностика. Методы: Пер. с англ./ Под ред. С. Херрингтона, Дж. Макги. – М.: 1999. – 558 с. 3. Шлегель Г. Общая микробиология: Пер. с нем. - М.: Мир, 1987. – 567 с. 4. Barrett J. F., Hoch J. A. Two-Component Signal Transduction as a Target for Microbial Anti-Infective Therapy/ Antimicrob. Agents Chemother. – 1998. – Vol. 42, No 7. – P. 1529–1536. 5. Claverys J. P., Martin B. Bacterial ‘competence’ genes: signatures of active transformation,or only remnants?// Trends Microbiol. – 2003. – Vol.11, No.4. – P. 161 – 165. 6. Fabret C., Feher V. A., Hoch J. A. Two-component signal transduction in Bacillus subtilis: how one organism sees its world/ J. Bacteriol. – 1999. - Vol. 181, No. 7. - P. 1975–1983. 7. Fitzgerald J. R., Musser J.M. Evolutionary genomics of pathogenic bacteria// TRENDS in Microbiol. – 2001. – Vol. 9 No.11. – P. 547 – 553. 8. Hacker J., Kaper J.B. Pathogenicity islands and the evolution of microbes / Annu. Rev. Microbiol. - 2000. – Vol. 54. - P. 641–79. 9. Hueck C. J. Type III protein secretion systems in bacterial pathogens of animals and plants / Microbiology and molecular biology reviews. – 1998. - Vol. 62, No. 2. - P. 379–433. 10. Krawiec S., Riley M. Organization of the bacterial chromosome/ Microbiol. Rev. - 1990. - Vol. 54, No. 4p. – P. 502-539. 11. Miller M. B., Bassler B.L. Quorum sensing in bacteria / Annu. Rev. Microbiol. - 2001. – Vol. 55. – P. 165–99 12. Ochman H., Santos S.R. Eyeing bacterial genomes// Current Opin. Microbiol. - 2003. – Vol. 6. – P. 109 – 113. 13. Parsek M. R., Greenberg E. P. Acyl-homoserine lactone quorum sensing in gram-negative bacteria: a signaling mechanism involved in associations with higher organisms// PNAS. – 2000. – Vol. 97, № 16. – P. 8789–8793. 14. Short-Sequence DNA Repeats in Prokaryotic Genomes/ A. Belkum, S. Scherer, L. Alphen, H. Verbrugh // Microbiol. Molec. Boil. Rev. – 1998. – Vol. 62, No. 2. - P. 275–293. 15. Tang Y. W., Procop G.W., Persing D. H. Molecular diagnostics of infectious diseases/ Clin. Chemistry. – 1997. – Vol. 43. – P. 2021-2038. 16. Toussaint A., Merlin C. Mobile Elements as a Combination of Functional Modules// Plasmid. – 2002. - Vol. 47. – P. 26–35.
[1] Геномные мутации отсутствуют у бактерий, в связи с наличием у них только одной хромосомы [2] Для студентов, занимающихся в кружке по микробиологии [3] Пробой в микроэррей исследованиях называют известные ДНК, пептиды; образцом, или мишенью называют изучаемую ДНК [4] Идентификацию секвенированных ДНК-фрагментов проводятпутем их сравнения с международным банком геномов (доступ через сервер http://www. ncbi.nlm.nih.gov/blast/) с использованием программы BLAST Дата добавления: 2015-12-16 | Просмотры: 486 | Нарушение авторских прав |
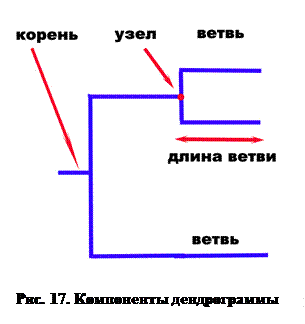 Ветви масштабированного древа калибруются для отражения количества произошедших изменений. Ветви немасшта-бированных деревьев могут содержать цифровую пометку о количестве произошедших изменений.
Ветви масштабированного древа калибруются для отражения количества произошедших изменений. Ветви немасшта-бированных деревьев могут содержать цифровую пометку о количестве произошедших изменений.